语言环境:R version 4.0.3
IDE:Pycharm
作图:Windows Excel
咋一看,好像不太理解什么是数据融合和重构。其实不仅在科研上,我们在日常工作中也无时无刻地都在接触数据整理,分析。现在我们从一个小例子出发,对数据融合与重构做一个全面解释。
图1 财务报表
一、概念引入
数据融合(melt)可以使得每一行都是唯一的标识符-变量组合。
数据重构(dcast)可以将融合后的数据重新构建自己想要的组合,包括函数计算。
二、数据融合
我所在实验室是从事小麦抗叶锈病基因定位、克隆与机理分析。下面从一个简单小实验就上述概念进行解释。注:数据已脱敏。
- 材料与方法
- 材料:抗病品种Sample,感病品种(对照)Control
- 方法:2种处理,2次重复(生物学实验常3-4次重复)
表1 实验数据
- 数据说明
- Treatment列:处理1和处理2
- Rep.列:重复1,重复2
- Sample列:抗病品种每次取样数据
- Control列:感病品种(对照)每次取样数据
- 融合操作
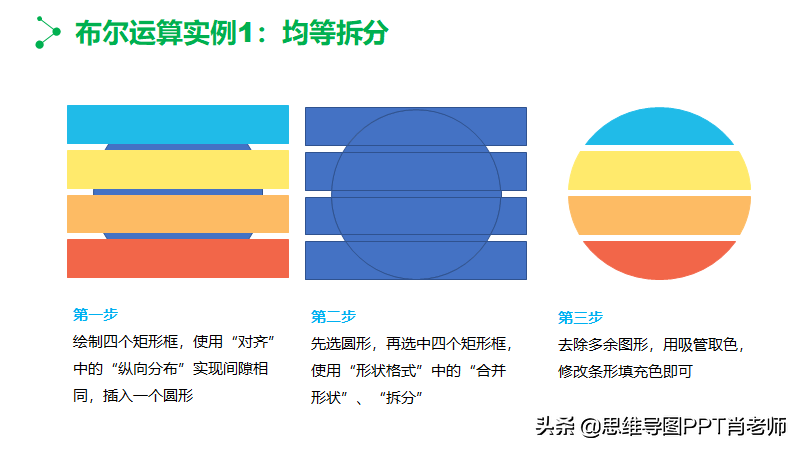
- 需求:将表1数据集整理成每行为单一的标识符-变量组合,即每个处理,重复,样品对应观测值。(表2)
表2 数据融合
- 实现:使用R语言reshape2包
图2 R语言
reshape2包是reshape包的升级版,功能更加强大。它是一套重构和融合数据集的万能工具。它并未包含在R的标准安装中,所以在使用时需要进行安装。
- 首先安装reshape2包并构建原始数据框,R脚本如下
install.packages("reshape2")
library(reshape2)
Treatment <- c(1,1,2,2)
Rep. <- c(1,2,1,2)
Sample <- c(8,7,2,2)
Control <- c(9,8,3,2)
mydata <- data.frame(Treatment,Rep.,Sample,Control)
Treatment Rep. Sample Control
1 1 8 9
1 2 7 8
2 1 2 3
2 2 2 2
- 重构原始数据mydata并赋值与新变量mydata2
mydata2 <- melt(mydata,id=c("Treatment","Rep."))
Treatment Rep. variable value
1 1 Sample 8
1 2 Sample 7
2 1 Sample 2
2 2 Sample 2
1 1 Control 9
1 2 Control 8
2 1 Control 3
2 2 Control 2
需要注意的是,重构时必须要指定每个测量所需的变量(Treatment,Rep.),表1中的变量名的变量(Sample,Control)由程序自动创建。对应列(variable,value)。
三、数据重构(重铸)
dcas()函数能够读取已经融合的数据,并根据提供的公式和函数将其进行重塑。调用格式如下:
newdata <- dcast(mydata2, formula, fun.aggregate)
代码说明:
mydata2为已融合数据,formula描述想要的最后结果,fun.aggregate(可选)为数据整合函数。
执行整合:
- 重构1
获取不同处理间抗病样品和感病对照观测值的平均值。
表3 重构1
newdata <- dcast(mydata2,Treatment~variable,mean)
Treatment Sample Control
1 7.5 8.5
2 2.0 2.5
- 重构2
获取不同重复间抗病样品和感病对照观测值的平均值。
表4 重构2
newdata <- dcast(mydata2,Rep.~variable,mean)
Rep. Sample Control
1 5.0 6
2 4.5 5
不执行整合:
- 重构3
进行数据调整
表5 重构3
newdata <- dcast(mydata2,Treatment+variable~Rep.)
Treatment variable 1 2
1 Sample 8 7
1 Control 9 8
2 Sample 2 2
2 Control 3 2
- 重构4
进行数据调整
表6 重构4
newdata <- dcast(mydata2,Treatment~variable+Rep.)
Treatment Sample_1 Sample_2 Control_1 Control_2
1 8 7 9 8
2 2 2 3 2
四、小结
因为重构1-2中指定了 mean 作为整合函数,所以对数据进行了重塑与整合。很多情况下,我们不得不在数据分析之前对其进行重塑或整合,那么这时候melt()和dcast()函数便推荐使用。
图3 数据表格